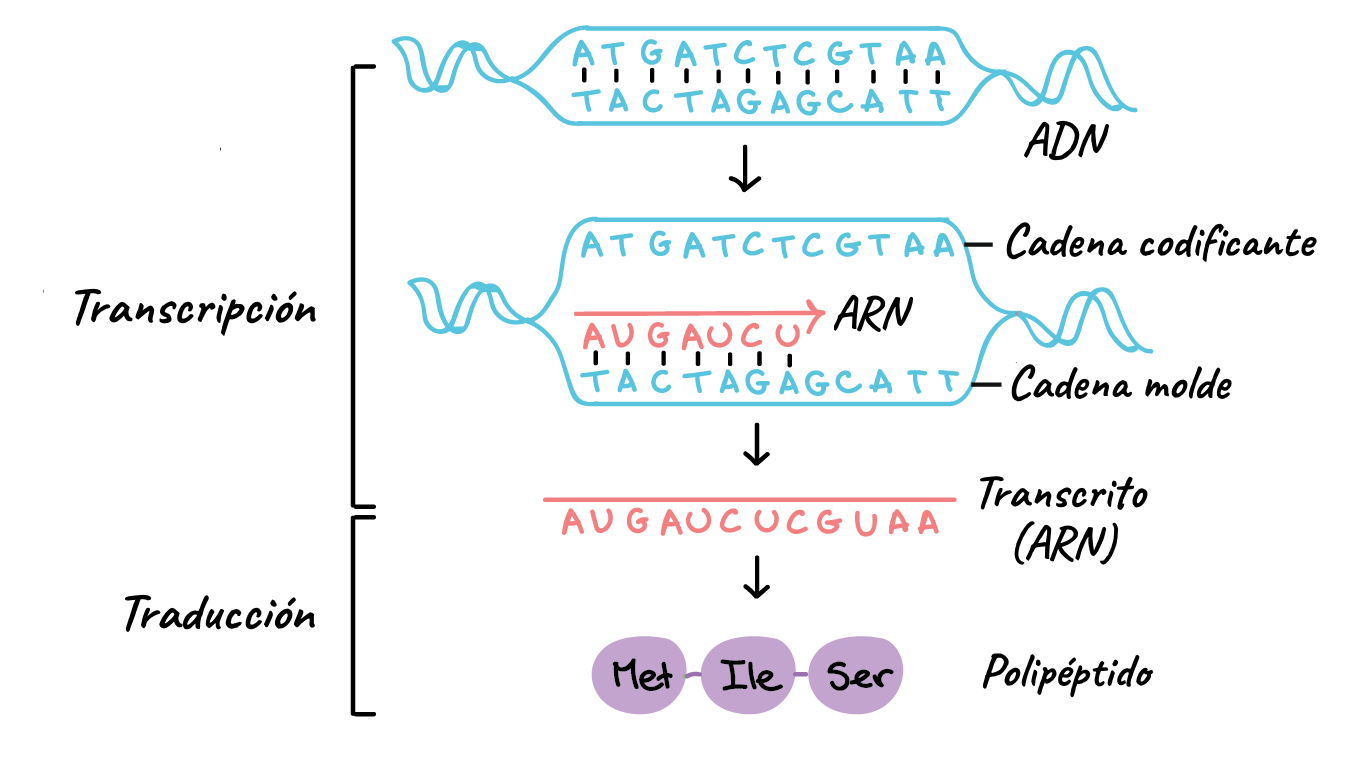
 Transcripción
TranscripciónLos genes son fragmentos de ADN que contienen la información necesaria para sintetizar una cadena peptídica: la información fluye del ADN al ARNm y a la proteína. Sin embargo, las funciones vitales (el metabolismo) son llevadas a cabo por proteínas.
Por esta razón, hay un mecanismo por el cual los genes expresan su información en forma de proteínas, la transcripción.
Pero, ¿cómo se expresan los genes? Pues bien, la expresión genética consiste en el paso de ADN a proteína. Se da en dos fases: transcripción (síntesis de ARN) y traducción (síntesis de proteínas).
- Transcripción. En este proceso una cadena del ADN de un gen hace de molde para sintetizar el ARN complementario. Aunque no todo el ADN se expresa, solo los genes que tengan información para formar una cadena peptídica, pasarán por la transcripción hasta llegar a ARNm y después por la traducción hasta llegar a proteínas. Hay genes que solo se transcriben y no se traducen, los que sintetizan ARNt y ARNr. Hay incluso secuencias de genes que ni se transcriben ni se traducen los que indican el comienzo y el fin de la transcripción etc.
- Traducción. La información que contiene el ARNm transcrito, pasará a ser traducido en los ribosomas, según el código genético.
Transcripción en Eucariotas
La transcripción es la primera fase de la expresión génica y consiste en transferir la información contenida en el ADN hasta el ARN.
En eucariotas, los ARN se sintetizan en el núcleo, después salen al citoplasma por los poros nucleares, se unen a los ribosomas y contribuyen a la traducción. Mitocondrias y cloroplastos también realizan transcripción y traducción.
La mayor parte de la transcripción consiste en sintetizar las moléculas de ARNm, para lo cual se necesita:
- Una cadena de ADN molde, de la cual solo se transcribe una cadena, la hebra molde, que tiene secuencias que son reconocidas por las enzimas que intervienen (secuencias promotoras). La dirección de lectura de la hebra molde es 3'-> 5' y el ARNm se sintetiza en dirección 5'->3'.
- Ribonucleótidos trifosfato de A, G, C y U, que deben estar en su forma trifosfato (ATP, GTP, CTP Y UTP), que les da energía para su esterificación. El ARNm se sintetizará mediante la unión, por enlaces éster, entre el fosfórico en 5’ de cada nuevo nucleótido con el -OH en 3’ del último nucleótido de la cadena.
- La enzima ARN polimerasa II, que se encarga de sintetizar el ARNm complementario de la hebra molde. Se forma mediante este proceso:
- Se reconoce las secuencias promotoras que indican el comienzo de la transcripción y usa los factores de transcripción
- Se recorre la hebra molde en dirección 3'->5' reconociendo su secuencia de bases y colocando los ribonucleótidos de A, G, C y U complementarios.
- Se cataliza la formación de enlaces éster entre los ribonucleótidos. Si el ribonucleótido trifosfato escogido es el complementario de la hebra molde, la enzima lo hidroliza, separando un resto pirofosfato y dejando un ribonucleótido monofosfato, que se esterifica con la cadena de ARNm gracias a la energía de dicha hidrólisis.
- Se reconoce las secuencias de terminación de la hebra molde. El proceso consta de tres fases: iniciación, elongación y terminación, seguidas de una maduración postranscripcional del ARNm.
- La iniciación. Para asegurarse de la eficacia y precisión de la transcripción hay tres clases de secuencias reguladoras en los genes: el promotor, que es la secuencia más cercana al inicio de la transcripción a los cuales se unen los factores de transcripción formando los factores basales; las secuencias potenciadoras, que están mucho más lejos y se unen a ellas los factores activadores de la transcripción que desempaquetan la cromatina y facilitan la unión de los factores basales con la ARN pol II, a través de los coactivadores; y las secuencias silenciadoras, que se encuentran entre las potenciadoras, y a ellas se unen los factores represores de la transcripción, que impiden la unión de los activadores a los potenciadores, reduciendo la velocidad de transcripción.
- La elongación. En el que la ARN pol II recorre la hebra molde en dirección 3'->5' y sintetiza ARNm en dirección 5'->3'.
- La terminación. En la que la ARN pol II reconoce la secuencia TTATTT de la señal de poliadenilación al final del último exón, la cual determina el fin de la transcripción y la separación del pre-ARNm de la cadena molde de ADN.

La maduración es la fase final donde se modifican los extremos del ARNm, se elimina los intrones y se alteran algunas secuencias en ciertos ARNm.
- Se añade en 5' una "caperuza" de metil-guanosina trifosfato, proceso que se realiza poco después de iniciar la transcripción, y que servirá de protección para el pre-ARNm de las nucleasas, así como reconocimiento del inicio de la traducción por los ribosomas.
- Se añade en 3' una "cola" de poli-adenina, mediante la actuación de la enzima poli-A polimerasa, que protegerá al ARNm.
- Se cortan los intrones y se pegan los exones, los intrones no serán traducidos por lo que tendrán que ser eliminados. Para ello, se produce un proceso del pre-ARNm que hace cortes entre intrones y exones. Después las secuencias intrónicas se enrollan como lazos y se eliminan, mientras que las exónicas se empalman, formando finalmente un ARNm funcional, que se unirá a los ribosomas para la traducción. Este proceso lo realizan ribonucleoproteínas pequeñas nucleares (RNPsn).
- Se edita o corrige el pre-ARNm, se introducen, eliminan o modifican (desaminación) ciertos nucleótidos del pre-ARNm.
 Es el paso de la información que contiene el ARNm (codificada como nucleótidos) a proteínas (codificada como aminoácidos). Para realizar el paso se utiliza el código genético que es una correspondencia entre nucleótidos y aminoácidos, que permite pasar el mensaje contenido en los genes hasta las proteínas. Como no existe ninguna relación química entre los tripletes de bases o codones y los aminoácidos, y, por tanto, los aa no se pueden unir directamente al ARNm hay unas moléculas (las de ARNt) que "traducen" de un lenguaje al otro. Las principales características del código genético son:
Es el paso de la información que contiene el ARNm (codificada como nucleótidos) a proteínas (codificada como aminoácidos). Para realizar el paso se utiliza el código genético que es una correspondencia entre nucleótidos y aminoácidos, que permite pasar el mensaje contenido en los genes hasta las proteínas. Como no existe ninguna relación química entre los tripletes de bases o codones y los aminoácidos, y, por tanto, los aa no se pueden unir directamente al ARNm hay unas moléculas (las de ARNt) que "traducen" de un lenguaje al otro. Las principales características del código genético son:- Es universal y no es ambiguo.
- Es degenerado o redundante. Cada aa puede estar codificado por más de un codón (codones sinónimos)
- Todos los codones tienen sentido, 5'->3'
- Carece de solapamiento, los codones no comparten bases entre sí y se colocan uno a continuación de otro.
- Primera adaptación: los ARNt son muy específicos y solo se pueden unir a un determinado aa. Para ello, utiliza la enzima aminoacil ARNt sintetasa, que tiene dos sitios activos específicos, uno que reconoce al aa y otro que reconoce al bucle o brazo D del ARNt.
- Segunda adaptación: se produce en los ribosomas. Los ARNt reconocen los codones del ARNm y se unen a ellos mediante su anticodón correspondiente y los ribosomas sirven de adaptador. La causa de la degeneración del código se denomina balanceo y es causada por la tercera base, la del extremo 5’, que puede unirse a diferentes bases. Los ARNt isoaceptores son los ARNt que aceptan el mismo aa.
La traducción consta de 3 etapas: iniciación, elongación y terminación
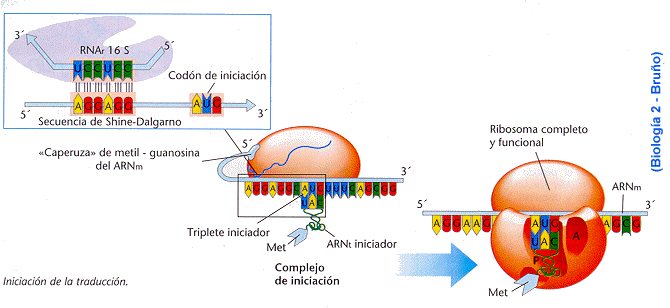
- Iniciación. Etapa en la que se forma el complejo de iniciación 80S, formado por un ribosoma unido al ARNm y al ARNt iniciador cargado con metionina. Tiene varias fases:

- Los factores de iniciación (FI) y la energía del GTP permiten la unión de la subunidad pequeña del ribosoma (40S) con el ARNt iniciador cargado con metionina.
- Este complejo reconoce la "caperuza" de metil guanosina trifosfato del ARNm y se une al extremo 5'.
- Con la energía del ATP, la subunidad pequeña del ribosoma se desplaza por el ARNm en dirección 5'-> 3' hasta que encuentra la primera secuencia AUG, que es el codón de iniciación. El ARNt iniciador cargado con metionina se une entonces a dicho codón.
- Se liberan los FI, se acopla la subunidad mayor (que tiene tres sitios de fijación: el E, el P y el A), y se forma el definitivo complejo de iniciación 80S.
- Elongación. Etapa en la que se sintetiza la cadena peptídica en el ribosoma, catalizado por la enzima peptidil transferasa. Consta de tres fases:
- Primera fase: entrada del ARNt-aminoácido. El sitio A es ocupado por el ARNt cuyo anticodón es complementario del siguiente triplete en el ARNm. Esto se consigue con el factor de elongación FE-1 y energía del GTP.
- Segunda fase: formación del enlace peptídico. La peptidil transferasa rompe el enlace de la metionina unida al ARNt iniciador cargado con metionina del sitio P y la une, mediante enlace peptídico, al que transporta el ARNt del sitio A. A este proceso ayudan las ribozimas del ribosoma.
- Tercera fase: traslocación. El factor de elongación FE-2, con energía del GTP, mueve el ribosoma a lo largo del ARNm, tres nucleótidos en dirección 5'-> 3'. Así el sitio P queda ocupado por el ARNt con el dipéptido, el sitio A queda vacío y el sitio E queda ocupado por el ARNti ya sin aa.
- Repetición de las tres fases: la entrada de un nuevo ARNtaa en el sitio A expulsa al ARNt del sitio E y se repiten otra vez las tres fases. Así, se va formando un tripéptido, luego un tetrapéptido, etc.
- Terminación. Etapa que tiene lugar cuando tras una traslocación aparece en el sitio A uno de los tres codones de terminación: UAA, UAG o UGA. Un factor proteico de terminación (RF) se une a dicho codón e impide la unión de un aminoacil-ARNt en el sitio A. La enzima peptidil transferasa cataliza entonces la transferencia de la cadena peptídica a una molécula de agua, lo que provoca la hidrólisis entre la cadena peptídica y el ARNt que lo portaba. La cadena peptídica queda así liberada; las dos unidades del ribosoma se separan y el ARNm puede volver a traducirse o es degradado.
- Plegamientos de las proteínas, para lograr estructuras más complejas.
- Adición de grupos prostéticos, como son glúcidos, lípidos etc.
- Modificación de algunos aa, por procesos como la fosforilación.
- Cortes proteolíticos, por los que se eliminan algunos aa de la cadena.
- En procariotas, la regulación se da principalmente en la transcripción y utilizan proteínas reguladoras codificadas con genes reguladores que pueden actuar como controles negativos, reprimiendo la transcripción o como controles positivos, intensificándola. Jacob, Monod y otros establecieron en modelo genético del operón (que es un grupo de genes estructurales cuya expresión está regulada por los mismos elementos de control y genes reguladores) que permite comprender cómo tiene lugar la regulación de la expresión génica en bacterias. Los principales elementos que constituyen un operón son los siguientes:
- Los genes estructurales: llevan información para polipéptidos y son genes cuya expresión está regulada. Los operones bacterianos suelen contener varios genes estructurales.
- El promotor (P): es un elemento de control que es una región del ADN con una secuencia que es reconocida por la ARN polimerasa para comenzar la transcripción y se encuentra justo antes de los genes estructurales.
- El operador (O): es otro elemento de control que es una región del ADN con una secuencia que es reconocida por la proteína reguladora y que se sitúa entre la región promotora y los genes estructurales
- El gen regulador (i): es una secuencia de ADN que codifica para la proteína reguladora que reconoce la secuencia de la región del operador y está cerca de los genes estructurales del operón pero no está justo al lado.
- Proteína reguladora: es una proteína codificada por el gen regulador que se une a la región del operador.
- Inductor: es un sustrato o compuesto cuya presencia induce la expresión de los genes.
Los genes estructurales del operón lactosa (necesarios para degradar la lactosa) codifican para la β-galactosidasa, que cataliza la hidrólisis de la lactosa en glucosa más galactosa; la galactósido permeasa, que transporta galactósidos al interior de la célula bacteriana; y una acetiltransferasa, que impide que se metabolicen otros galactósidos que no sean la lactosa.
Si no hubiera inductor (la lactosa), la proteína represora, se unirá a la región operadora e impedirá la unión de la ARN-polimerasa a la región promotora y no se transcribirán los genes estructurales.
Si hay inductor (la lactosa), se unirá a la proteína reguladora que cambia su conformación y se suelta de la región operadora, dejando acceso a la ARN-polimerasa para que se una a la región promotora y se transcriban los genes estructurales. La presencia del inductor hace que se expresen los genes estructurales del operón, necesarios para metabolizar la lactosa.
- En eucariotas, la regulación de la expresión génica es más compleja y menos conocida. Tiene lugar en cinco niveles:
- Estructura de la cromatina, que se controla mediante metilación del ADN, metilación de histonas y acetilación de histonas.
- Control de la transcripción, mediante elementos traslocables o genes saltarines, como transposones y retrotransposones, que pueden inactivar o sobreactivar la transcripción; y mediante los factores de transcripción (potenciadores o enhancers e inhibidores o silencers).
- Control de la maduración postranscripcional.
- Control de la traducción: degradación de los ARNm, actuación de ARNi (ARN de interferencia), etc.
- Control del proceso postraduccional: maduración, cambios conformacionales, degradación por el proteasoma, etc.



Comentarios
Publicar un comentario
Gracias por comentar. Te rogamos que seas preciso y educado en tus comentarios.